Proteine
(Eiweiße, Eiweißstoffe, Eiweißkörper). Auf Berzelius zurückgehende u. seit Mulder (1838) gebräuchliche u. von griech.: proteuein = „der Erste sein“ abgeleitete Sammelbez. für natürlich vorkommende Copolymere, die sich in der Regel aus 20 verschiedenen a-Aminosäuren (im folgenden: AS) als Monomeren zusammensetzen. Von den nahe verwandten Polypeptiden werden sie aufgrund ihrer mol. Größe unterschieden, wenn auch nicht immer streng abgegrenzt: Ab etwa 100 Monomer-Einheiten (AS-Resten) spricht man meist von Proteinen. Es ergeben sich MR von 10 000 bis mehrere Millionen. siehe Artikel Proteine von Franci & Kathi
Peptide
(von griech.: peptos = verdaulich). Bez. für durch Peptid-Bindungen Säureamid-artig verknüpfte Kondensationsprodukte von Aminosäuren. Siehe Artikel Aminosäuren von Jana und Imke Spö.
Abb.: Allg. Strukturformel der Peptide. Bauen sich die Mol. aus 2 Aminosäure-Resten auf, so spricht man von Dipeptiden, bei 3 u. mehr von Tri-, Tetra-, Pentapeptiden etc.; P. mit 2–10 Aminosäure-Resten faßt man als Oligopeptide, solche mit 10–100 als Polypeptide zusammen, doch ist der Übergang von den letzteren zu den höhermol. Proteinen (Eiweißstoffen) nicht genau definiert. P. mit Bindungen zwischen den seitenständigen Amino-Gruppen von Diaminocarbonsäuren (z. B. Lys) u. seitenständigen Carboxy-Gruppen von Aminodicarbonsäuren (z. B. Glu, Asp) statt der üblichen Peptid-Bindungen zwischen a-NH2 u. -COOH nennt man Isopeptide; die von mehrfunktionellen Aminosäuren wie Glu, Asp, Lys, Arg u. Desmosin ausgehenden zusätzlichen Bindungen sind für die Entstehung von P.-Netzstrukturen verantwortlich. P., deren Aminosäure-Sequenz relativ zu einem bestimmten anderen P. die gegenläufige Reihenfolge an Aminosäuren aufweisen, werden als Retropeptide bezeichnet. Zur Schreibung von P.-Formeln benutzt man meist 1- od. 3-Buchstaben-Notationen für die Aminosäuren, s. die Liste dort. Z. B. stehen AG od. Ala-Gly für L-Alanylglycin [H2N–CH(CH3)–CO–NH–CH2–COOH] u. GA od. Gly-Ala für isomeres Glycyl-L-alanin [H2N–CH2–CO–NH–CH(CH3)–COOH]; falls nicht anders gekennzeichnet (etwa durch: Gly¬Ala), steht links die (freie od. protonierte) Amino-Gruppe u. rechts die (freie od. deprotonierte) Carboxy-Gruppe.
Biolog. Bedeutung: Auf die Bedeutung der makromol. P. für pflanzliche u. tier. Organismen wird bei Proteine ausführlich eingegangen. Eine gleichermaßen spezif. Rolle spielen Oligo- u. Polypeptide im tier. Organismus z. B. als Hormone (Peptidhormone), Wachstumsfaktoren, Cytokine, Neurotransmitter u. Neuromodulatoren (Neuropeptide). Für die physiolog. Wirkung der P. ist neben der Konfiguration die Konformation u. die mol. Dynamik von Bedeutung, u. natürlich benötigen die P., um als Mediatoren wirksam werden zu können, spezif. Rezeptoren. Bei der Zell-vermittelten Immunantwort werden Antigene (Fremd-Proteine) von Antigen-präsentierenden Zellen zu P. (Antigen-Peptide, T-Zell-Epitope) abgebaut, von Histokompatibilitäts-Antigenen komplexiert u. so an der Zelloberfläche den T-Lymphocyten zum „Abtasten“ dargeboten; von außen verabreichte P. (peptide feeding) werden ebenfalls präsentiert. Auch von körpereigenen Proteinen abgeleitete Selbst-P. werden präsentiert, was in der Frühphase der T-Zell-Entwicklung für die Entstehung von Selbst-Toleranz von Bedeutung ist. P.-Ester können für süßen (Aspartame®) od. bitteren Geschmack verantwortlich sein, u. wieder andere P. treten als Toxine pflanzlichen od. tier. Ursprungs in Erscheinung. Auch unter den Antibiotika finden sich eine Reihe von P. (Peptid-Antibiotika ), die z. T. Aminosäuren der „unnatürlichen“ D-Konfiguration enthalten, ggf. auch Hydroxycarbonsäuren, die über Esterbindungen verknüpft sind (Peptolide). Viele der physiolog. aktiven P. liegen als homodete od. heterodete Cyclopeptide vor.
Herst.: Auch bei der Synth. ist der zeitliche Aufwand aufgrund der Entwicklung automat. Verf. u. der Festphasen-Technik (Merrifield-Technik) ungleich geringer geworden. Für die Herst. biolog. aktiver u. pharmakolog. nutzbarer P. werden heute neben der chem. Peptid-Synthese in zunehmendem Maße Meth. der Biotechnologie u. Gentechnologie eingesetzt, was z. B. auf dem Gebiet der Peptidhormone bereits zu Erfolgen geführt hat.
Biosynth.: Meist durch enzymat. „Resektion“ aus Proteinen, die nach Maßgabe des genetischen Codes (Näheres s. dort) u. der Sequenzinformation der Messenger-Ribonucleinsäuren in den Ribosomen gebildet werden, vgl. Peptidhormone. In manchen Fällen findet jedoch durch nicht-ribosomale Enzyme eine Biosynth. von P. aus den Aminosäuren statt .
Peptidbinung
Eine Peptidbindung (-NH-CO-) ist eine Bindung zwischen der Carboxylgruppe einer und der Aminogruppe einer zweiten Aminosäure.
Zwei Aminosäuren können (formal) unter Wasserabspaltung zu einem Dipeptid kondensieren.
Im Beispiel reagieren zwei Moleküle der einfachsten Aminosäure Glycin zu einem Dipeptid: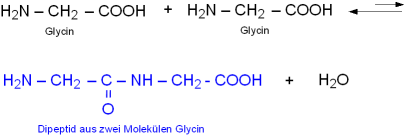
Struktur der Peptidgruppe
- Bestiummung der Bildungslänge in Peptidbindung durch Röntgenstrukturanalyse.
- CN-Bindung ist kürzer als bei Aminen
- durch die Delokalisierung der Peptidgruppe ist ein besonders stabil Verbindungsverhältnis erreicht.
Aufgabe
Glutathion ist ein Tripeptid mit der Aminosäuresequenz (Glu-Cys-Gly). Es schützt in lebenden Zellen Verbindungen vor Oxidation.Dabei wird Glutathion selbst oxidiert. (m(Glutathion)=307 g x mol-1;M(Oxidationsprodukt)= 612g x mol-1) a)Geben Sie die Strukturformel des Tripeptids als Zwitterion an, beachten Sie dabei, das Glutaminsäure im Glutathion eine y-Peptidbindung ausbildet b)Leiten Sie die Strukturformel des Oxidationsproduktes aus Angaben der molaren Masse ab c)Geben Sie die Reaktionsgleichung der Oxidation mit Hilfe der Strukturformelen an. Bennen Sie den entstehenden Bindungstyp.
Quellen
Chemie heute Seite 375 Kapitel 19.8,Römpp Lexikon Chemie – Version 2.0, Stuttgart/New York: Georg Thieme Verlag 1999
